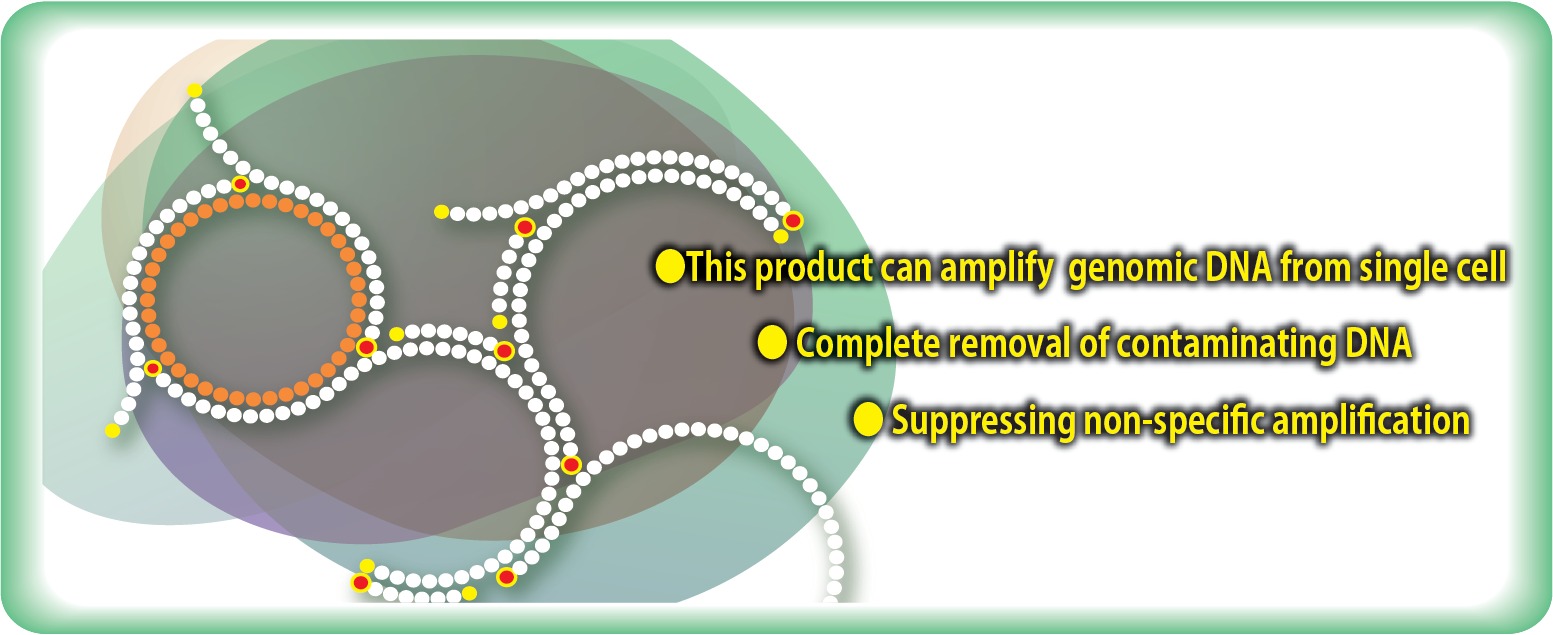
WGA 반응 설정 조건 및 제품 권장사항 (phi29, WGA, T7 Endonuclease I 활용)
Whole Genome Amplification (WGA)은 극미량의 DNA를 증폭할 때 필수적인 기술입니다. 특히 phi29 DNA polymerase를 사용하면 높은 신장성(processivity)과 오류율이 낮은 증폭이 가능해집니다. 이번 포스팅에서는 효율적인 WGA 반응 조건, 최적화된 시약 조합, 그리고 시퀀싱 전 필수적인 T7 Endonuclease I 처리 방법까지 상세히 다뤄보겠습니다.
1. WGA 반응의 핵심: phi29 DNA polymerase
phi29 DNA polymerase는 rolling circle amplification (RCA)과 WGA에 최적화된 효소입니다. 기존의 Taq polymerase와 비교했을 때 3′→5′ exonuclease 활성(proofreading)이 있어 오류가 적고, 긴 DNA 조각(>70 kb)도 안정적으로 증폭할 수 있습니다.
✔ phi29를 사용하는 이유
- 높은 processivity: 한 번 결합하면 수십 kb를 연속적으로 합성합니다.
- 저오류율: PCR보다 돌연변이 발생 확률이 현저히 낮습니다.
- Strand displacement 활성: 추가적인 변성 단계 없이도 이중가닥 DNA를 풀어냅니다.
“그렇다면 정확히 어떤 조건에서 반응을 설정해야 할까요?”
2. WGA 반응 조건 상세 가이드
2-1. 기본 반응 구성
| 시약 | 부피 (μl) | 최종 농도 |
|---|---|---|
| 10X phi29 Buffer | 2.5 | 1X |
| Random hexamer primer* | 2.5 | 50 μM |
| dNTP (10 mM) | 2.5 | 1 mM |
| DNA (genomic or plasmid) | 변수 | 10 pg–1 ng/반응 |
| BSA (20 mg/ml) | 0.125 | 0.1 mg/ml |
| phi29 polymerase | 1 | 0.4 units/μl |
| Nuclease-free water | 총 25 μl | – |
📌 주의사항
- Primer: Exo-resistant random hexamer (3′ 말단에 phosphorothioate 2개 포함) 사용
- DNA 양: WGA는 10 pg 이상, RCA는 100 fg plasmid DNA부터 가능
2-2. 선택적 열변성(Heat Denaturation) 단계
일부 프로토콜에서는 95°C, 3분 변성을 권장하지만, phi29의 strand displacement 능력으로 생략 가능합니다. 다만, GC-rich DNA나 복잡한 구조의 경우 변성 단계를 추가하면 수율이 향상될 수 있습니다.
✔ 반응 순서
- Phi29, BSA를 제외한 모든 시약을 혼합
- 95°C, 3분 → 실온 냉각 (변성 시 사용)
- BSA + phi29 추가 후 30°C, 2시간 이상 반응
- 65°C, 10분으로 효소 불활성화
3. WGA 산물 정제 및 고수율을 위한 팁
3-1. SPRI beads를 이용한 정제
증폭 후 0.8X SPRI beads로 정제하면 primer, dNTP, 효소 잔여물을 제거할 수 있습니다.
3-2. Inorganic Pyrophosphatase (IPP)로 수율 향상
반응 중 생성되는 pyrophosphate (PPi)는 효소 활성을 저해할 수 있습니다. 1 μl IPP (M0361)를 추가하면 증폭 효율이 개선됩니다.
“그런데 시퀀싱을 할 계획이라면?”
4. 시퀀싱 전 필수 단계: T7 Endonuclease I 처리
WGA 산물은 hyperbranched 구조를 형성하기 때문에, Nanopore 또는 Illumina 시퀀싱 시 로딩 효율이 급격히 떨어집니다. T7 Endonuclease I로 분기를 절단하면 더 깨끗한 데이터를 얻을 수 있습니다.
✔ De-branching 반응 조건
| 시약 | 부피 (μl) | 최종 농도 |
|---|---|---|
| WGA 산물 | 변수 | – |
| 10X NEBuffer 2 | 3 | 1X |
| T7 Endonuclease I | 1.5 | 0.5 units/μl |
| Nuclease-free water | 총 30 μl | – |
37°C, 30분 반응 후 SPRI beads로 정제하면 시퀀싱에 최적화된 DNA를 얻을 수 있습니다.
5. 자주 묻는 질문 (FAQ)
Q1. WGA와 PCR의 차이점은?
- PCR: 특정 부위만 증폭, 높은 오류율, 짧은 산물 (~10 kb)
- WGA (phi29): 전체 게놈 증폭, 낮은 오류율, 긴 산물 (>70 kb)
Q2. DNA 양이 10 pg 미만이면 증폭이 안 될까요?
Single-cell WGA도 가능하지만, primer 최적화와 BSA 추가가 필수적입니다.
Q3. T7 Endonuclease I 처리 후 전기영동으로 확인할 수 있나요?
Hyperbranched DNA는 gel에서 smear로 보이지만, T7 처리 후 뚜렷한 band가 관찰됩니다.
6. 마치며
phi29 기반 WGA는 미량 DNA 증폭부터 단일 세포 시퀀싱까지 다양한 연구에 활용됩니다. 열변성 단계 최적화, T7 Endonuclease I 처리만 잘해도 데이터 품질이 크게 달라집니다. 이 가이드가 여러분의 실험에 도움이 되길 바랍니다!
“더 궁금한 점이 있다면 댓글로 남겨주세요!”